


近日,湖南农业大学水产学院李德亮和李军华教授团队与湖南省水产研究所宋锐研究员团队的合作研究通过高通量定量PCR和16S rRNA基因测序,阐明了湖南省内湘江流域的水体理化性质以及鱼类肠道ARG图谱和微生物群落分布,研究成果以“Comparative analysis of antibiotic resistance gene (ARG) profiles and microbial communities in the Xiangjiang River highlights fish gut opportunistic pathogens as potential reservoir of high-risk ARGs”为题发表于Water Biology and Security(IF 5.1)。
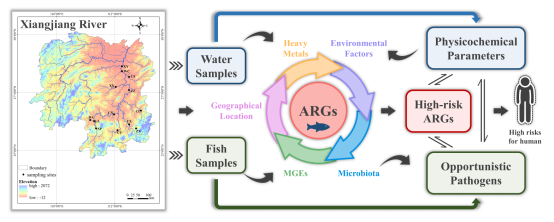
抗生素抗性基因(Antibiotic resistance genes, ARGs)是新兴的环境污染物,其扩散和传播已成为全球公共卫生面临的重大挑战。目前,关于自然水域生态系统中ARGs分布特征与传播机制的研究有限。该研究基于高通量定量PCR和16S rRNA基因测序的比较分析,系统地确定了湖南省内湘江流域鱼类肠道ARG图谱及微生物群落分布。研究结果表明,湘江鱼类肠道ARGs的组装以确定性过程主导,受到可移动遗传元件(mobile genetic elements, MGEs)、环境因素和微生物的影响。此外,湘江鱼类肠道ARGs在不同采样点的分布存在差异,以大环内酯类-林可酰胺类-链阳霉素类B(macrolide-lincosamide-streptogramin B, MLSB)和磺胺类为主,并且在湘乡(Xiangxiang, XX)的平均相对丰度最高,在茶陵(Chaling, CL)最低。值得注意的是,湘江鱼类肠道条件致病菌和高风险ARGs存在显著相关性,提示其可能为高风险ARGs的潜在存储库。研究结果为制定针对性的策略以管理和评估自然水域生态系统中的ARGs提供了宝贵见解。
湖南农业大学水产学院博士研究生徐杨和硕士陈睿为论文的共同第一作者,宋锐研究员、李德亮教授和李军华教授共同通讯作者。本研究得到了国家自然科学基金、湖南省自然科学基金杰出青年基金、湖南省教育厅重点研究项目和现代农业产业技术体系建设专项等项目的资助。
全文链接:https://doi.org/10.1016/j.watbs.2025.100444